Storia dell'articolo
Chiudi
Questo articolo è stato pubblicato il 10 marzo 2011 alle ore 06:52.
Quando, nel 1953, Watson e Crick individuarono la struttura a doppia elica del Dna, il ruolo di questa molecola nella trasmissione dei caratteri ereditari era già noto. I risultati di Avery, Leod e McCarthy nel 1944, confermati da quelli di Hershey e Chase nel 1952, avevano già chiaramente dimostrato che svolgeva un ruolo chiave nel trasportare informazioni dai genitori ai discendenti. Tuttavia lo scoprire che la molecola è un nastro sul quale le informazioni sono scritte in un alfabeto di soli quattro caratteri (le lettere A, C, G e T; cioè le basi azotate adenosina, citosina, guanina e timina) ha fatto subito sognare una tecnologia in grado di leggerle. Impresa difficile, dato che una base misura circa 0,33 nanometri, e in un singolo cromosoma umano possono essercene quasi 250 milioni.
I primi tentativi furono penosamente lenti. Tuttavia col tempo sono state sviluppate tecniche affidabili. Una delle più efficaci è quella dei terminatori di catena sviluppata da Frederick Sanger all'inizio degli anni 70. In questo caso il Dna viene sottoposto a reagenti che si sostituiscono alle basi, spezzando la molecola e marcandone i frammenti con elementi fluorescenti o radioattivi, diversi a seconda della base contenuta. Successive tecniche di analisi riescono a ricostruire con precisione l'ordine delle basi all'interno della molecola.
Le tecniche usate oggi sono dette "a elevato parallelismo", perché consentono di analizzare in parallelo molti spezzoni di Dna, operando in modo automatizzato e rapido. Per esempio, la tecnologia detta "ion semiconductor" utilizza un sensore in grado di percepire gli ioni idrogeno prodotti in occasione della polimerizzazione di un filamento di Dna. Sottoponendolo a reagenti diversi che producono ciascuno la polimerizzazione di una singola base, si può identificare la posizione di ogni base. Un'altra tecnica è quella delle nanosfere, che fa sì che la molecola di Dna si suddivida in una serie di agglomerati sferici, che possono essere disposti in una griglia e analizzati in parallelo.
Il vertiginoso calo dei tempi e dei costi necessari per sequenziare una molecola di Dna fa pensare che valga una regola empirica simile alla cosiddetta "legge di Moore" per la potenza di calcolo dei microchip. Solo che la rapidità di analisi del Dna cresce ancora più rapidamente. Il National Human Genome Research Institute di Bethesda ha calcolato i costi per sequenziare un intero genoma dal 2000 a oggi, evidenziandone la vertiginosa caduta.
Di recente un'equipe di scienziati dell'Imperial College di Londra ha annunciato sulla rivista «Nano Letters» di aver realizzato un chip in grado di analizzare interi filamenti di Dna. È dotato di un nanoporo del diametro di 50 nanometri, al cui interno c'è una coppia di elettrodi di platino. Il flusso di corrente viene modificato in modo diverso dal passaggio di ciascuna base. Il chip è così in grado di leggere in sequenza il contenuto di informazione di un filamento di Dna che lo attraversi. La tecnologia ancora non è stata provata su interi genomi, tuttavia gli autori ritengono che abbia la potenzialità di leggere milioni di basi al secondo. Essendo basata su chip il cui costo decresce enormemente con la produzione su grande scala, potrebbe portare in breve tempo l'analisi del Dna alla portata di chiunque.
Chi immagina un'epoca in cui manipolare il Dna sarà facilissimo deve però tenere conto che, se presto saremo in grado di leggere il linguaggio dei geni in modo rapido ed economico, ancora sappiamo ben poco su come interpretarlo. È ancora da vedere se, all'enorme massa di dati grezzi presto disponibili, corrisponderà un miglioramento altrettanto massiccio della nostra comprensione sul modo in cui dai geni può svilupparsi la complessità di un essere vivente.
© RIPRODUZIONE RISERVATA
Permalink
Ultimi di sezione
-
APP AND ENTERTAINMENT
Ecco le migliori app per organizzare le vacanze last-minute
di Anna Volpicelli
-
nova24
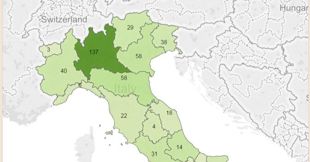
Startup, la mappa delle regioni che hanno ricevuto più finanziamenti dal Fondo Pmi
di Luca Tremolada
-
TREND
Effetto Grecia anche su Google: vacanze e crisi sono il tormento degli italiani
-

Gadget
I Google Glass cambiano faccia: arriva la Enterprise Edition?
-

DIGITAL IMAGING
Fotografia: ecco le migliori app per i professionisti dell'immagine - Foto
di Alessio Lana
-

la app della discordia
UberPop sospeso anche in Francia. E ora in Europa è fronte comune